La idea de este proyecto nació durante los primeros meses de la pandemia. En marzo y abril de 2020 los ingresos hospitalarios por COVID-19 generaron un problema de suministro de medicamentos, ante lo que se hizo necesario investigar en la literatura científica sobre la enfermedad para acudir a fármacos alternativos disponibles.
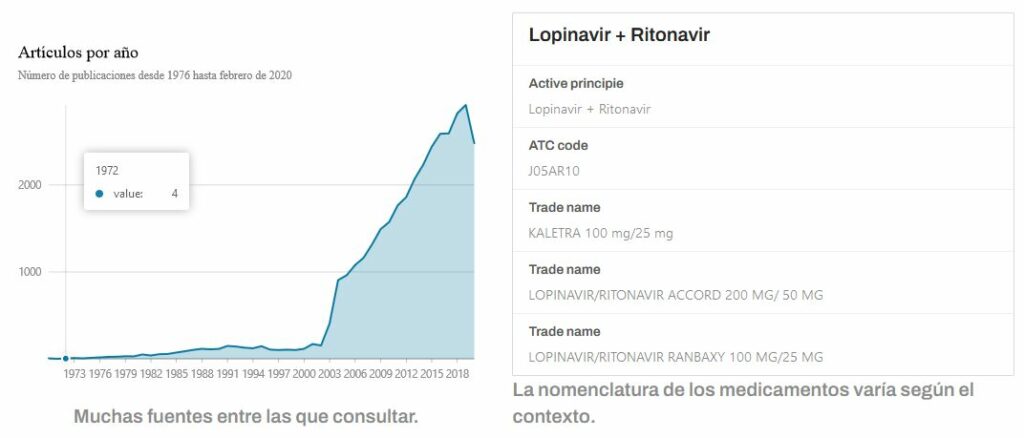
La literatura científica publicada, donde se describen los principios activos, los fármacos y su uso, se demostró tan numerosa como difícil de gestionar, hasta el punto de que la Oficina de Política Científica y Tecnológica de la Casa Blanca hizo un llamamiento a la comunidad de Inteligencia Artificial internacional, poniendo en abierto su repositorio CORD-19 sobre artículos científicos relacionados con el coronavirus. Se trata de un repositorio de más de 300.000 documentos científicos en inglés, de entre los cuales se dispone del texto completo de más de 200.000.
El Ontology Engineering Group de la Universidad Politécnica de Madrid, con una exitosa trayectoria en las áreas de Ingeniería Ontológica, Web Semántica, Datos Enlazados, Procesamiento de Lenguaje Natural y e-Ciencia Semántica, contribuyó lanzando el proyecto Drugs4Covid.
El corpus CORD-19 ha sido la principal base de datos utilizada en este estudio. Cada semana se publica una nueva actualización que incrementa la colección inicial con nuevas publicaciones. La edición de abril de 2020, con 60.702 artículos científicos y 2.103.891 párrafos, fue la primera que se utilizó en el estudio. La última actualización indexada es la edición de enero de 2022, con 334.580 artículos científicos y más de 10 millones de párrafos.
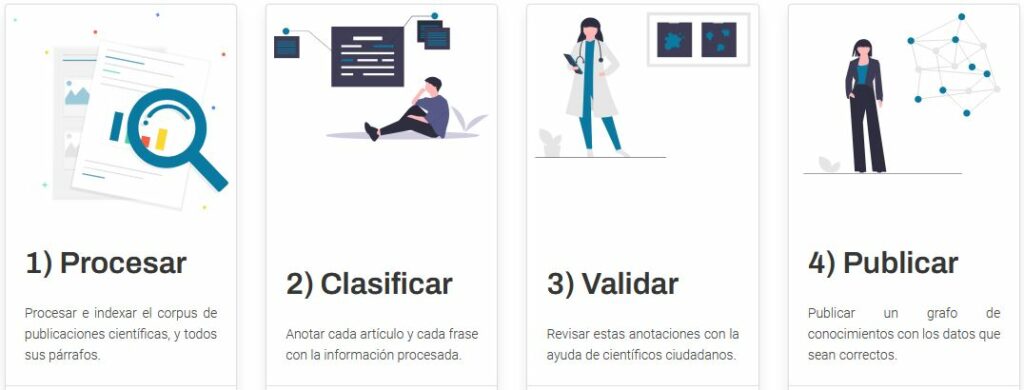
Todo el contenido está siendo analizado mediante técnicas de procesamiento de lenguaje natural y extracción de conocimiento a través de un proceso en 4 pasos: (1) procesando e indexando el corpus de artículos y párrafos mediante la identificación de los principios activos, grupos terapéuticos, síntomas y enfermedades. (2) Anotando automáticamente, usando técnicas de Inteligencia Artificial, cada texto con esta información, y pidiendo la ayuda de científicos ciudadanos para (3) revisar estas anotaciones y proponer relaciones entre ellas. Finalmente, (4) publicando un grafo de conocimientos con todos los datos y sus evidencias, esto es, conectando y unificando de manera significativa la información y haciéndola interrogable de manera natural a través de aplicaciones que faciliten la búsqueda y navegación. De cara a facilitar el acceso a la información recogida en el grafo de conocimientos, se está desarrollando una interfaz de pregunta-respuesta que permite realizar consultas en lenguaje natural y devuelve respuestas también en lenguaje natural.
Todos estos resultados se están publicando en forma de datos abiertos para que puedan ser utilizados por la comunidad sanitaria y por cualquier otro equipo de investigación que quiera resolver problemas adicionales. De hecho, los modelos de lenguaje que se han creado para identificar medicamentos, enfermedades y genes en la literatura científica tuvieron más de 100.000 descargas en HuggingFace en su primer mes de publicación.
Más información del proyecto en su web: https://drugs4covid.oeg.fi.upm.es/